CRISPR發展之英雄榜
德州大學分子生物科學研究所馬千惠編譯/臺大科教中心陳藹然責任編輯
編譯來源:Lander, E.S. (2016). The Heroes of CRISPR. Cell 164, 18-28.
2013年一篇指出 CRISPR (clustered regularly interspaced palindromic repeats) 技術可以準確且有效率地編輯活體真核細胞基因體的研究報告,在科學界颳起一陣風暴,從生物醫學到農業,有成千個實驗室應用此技術。本文將探討從二十年前起發現微生物的基因體含有奇怪的重複序列,經確認為此一適應性免疫系統,到了解其生物特性,進而運用在基因工程上的發展過程,以及這科學研究歷程給我們的啟示。
CRISPR 規則性間隔重複迴文序列群的發現,起源於西班牙一地中海海港,Francisco Mojica 的指導教授發現生活在沼澤中一耐鹽性極高的古細菌,其基因體會因為生長環境鹽度不一樣,而被限制酶切出不同片段。Mojica分析這些不同的基因片段,發現一個特殊結構含有類似迴文的重複序列及間隔,和其他已知的微生物重複序列不同。之後十年的時間,他致力於解開這結構的神密之處,發現其他的古細菌也具有相似的重複序列,而一日本研究團隊在細菌裏也發現類似的結構。Mojica認為如此相去甚遠的微生物卻有相似的結構,在原㧡生物中一定有其特殊意義。由於Mojica才剛開始教職工作,研究經費不足,只好轉而以生物資訊學來分析這奇特的重複序列,並命名為 SESRs (short regularly spaced repeats),之後修改成 CRISPR (clustered regularly interspaced palindromic repeats)。
20世紀結束時,Mojica已在二十種微生物基因體內找到 CRISPR序列。其他研究團隊在不同微生物也有相同的發現,並且找到CRISPR 相關基因(CRISPR associated genes, cas),但是其生物功能仍是未知,於是眾多假說被提出。後來Mojica 在分析重複序列之間的間隔序列(spacer) 有了重大發現: 一特定種大腸桿菌的 CRISPR 的間隔序列,居然和感染許多不同種大腸桿菌的P1 噬菌體的基因序列吻合,可是此一特定種的大腸桿菌本身並不會被P1 噬菌體感染,也就是説擁有此間隔序列,可以保護細菌本身不會再被同一噬菌體感染。他又繼續分析了4,500 段間隔序列,其中88段和已知序列吻合,其中三分之二是來自病毒或接合質體。可是當他要發表此重大的發現時卻連續被各大期刊拒絕,經過十八個月的努力,終於在2005年發表於 Journal of Molecular Evolution。
同時CRISPR系統的研究在世界各地陸續有不同的進展。法國國防部的科學家 Gilles Vergnaud 分析造成越南1964-1966年鼠疫的61株樣品,發現其重複序列都相同,只有其同事Christine Pourcel發現在CRISPR 的前端間隔序列 (spacers)有所不同,因此提出CRISPR可能是將過去受感染的記憶保存下來,保護細菌不再受感染,但是他們的論文同樣面對被期刊拒絕的狀況,最後晚 Mojica一個月的時間發表於 Microbiology。服務於法國一食品公司的研究者Philippe Horvath和其同事Rodolphe Barrangou在2007年以實驗證明 CRISPR 是一適應性防禦免疫機制,同時他們也發現 Cas7參與形成新的間隔和重複序列,以及 Cas9 具有兩種核苷酸酶部位HNH和 RuvC可以切斷核苷酸,且為達到完全的免疫功效,間隔序列和標的序列必須完全相同。
2008年德國科學家John van der Oost 和其同事首先直接設計出以CRISPR為基礎的免疫機制,像是幫細菌打了疫苗,不再受噬菌體感染。美國科學家 Luciano Marraffini和Erik Sontheimer 則證實 CRISPR 是以 DNA 為標的,並首先提出CRISPR 可能可以應用於不同物種的基因編輯,雖然有提出專利申請,但由於缺乏實驗佐證,後來終究放棄。
Sylvain Monieau 延續 Barrangou 先前對 Cas9的研究,發現 Cas9會準確地切在PAM(proto-spacer adjacent motif) 前三鹼基對的位置。另外 Emmanuelle Charpentier 和 Jὂrg Vogel團隊則發現在細胞內第三多的轉錄物 tracrRNA(trans-activating CRISPR RNA)會和由CRISPR序列所轉錄出來的crRNA前身結合,再由 RNaseIII修剪為具功能性的產物,研究至此階段已經能掌握CRISPR系統所需的要件。接下來是 Virginijus Siksnys成功地將 CRISPR 系統移至不同的生物體,達到一重要里程碑,確認 CRISPR-Cas9 系統所需且足夠的元件:Cas9 核苷酸䩈、crRNA以及tracrRNA。他們以純化的 Cas9蛋白研究整個過程的細節,最戲劇化的發現是利用設計CRISPR裏不同的間隔序列(spacer),讓此系統去辨識特定的基因位置並切斷 DNA,Siksnys 在2012年以此申請美國專利。大約在同時 Charpentier 和 Jennifer Doudna 合作也有同樣的發現和進展,他們更進一步將crRNA和tracrRNA 連結成sgRNA (single-guide RNA),此一概念後來被廣泛使用,經修改可以更有效地在生物體內作用。兩個團隊也同時都提出此一系統未來在生物科技上的應用潛力。
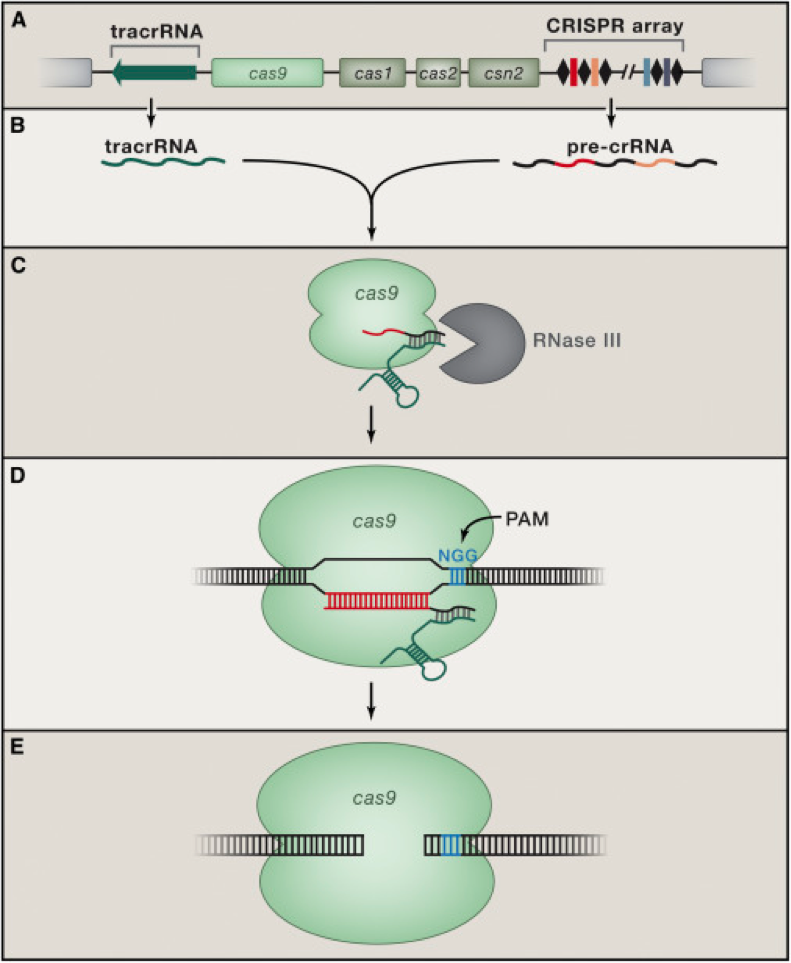
下一個重大的發展來自華裔科學家張鋒 (Feng Zhang),他進一步修改 Cas9,找到適合存在人類細胞內的 tracrRNA,成功地將CRISPR系統應用到哺乳動物細胞,可以高效率且準確地修改特定基因,張鋒更進一步以此系統做出複雜的老鼠系統來研究遺傳疾病及癌症,並且從全基因庫篩檢,研究特定生物過程的必需基因,同時他和生物資訊學者Kooin 發現新的 Class 2 CRISPR系統,只需另一核苷酸酶及 crRNA兩個元素便足夠。張鋒在2013年初於 Science 發表論文,成為此一領域被引用最多的文章。另一科學家 George Church也和張鋒有相同的發現,即以全長的crRNA-tracRNA連結在一起,在哺乳動物細胞內的功能比較好,他同時編輯七處基因,分析不同的基因重組機制,並在同一期期刊發表編輯人類基因體的論文。至此眾多科學家們陸續成功地以CRISPR為基礎去編輯許多不同生物的基因體,更朝人類基因療法及農業生產發展,甚至提出以此技術去設計人類胎兒的可行性。
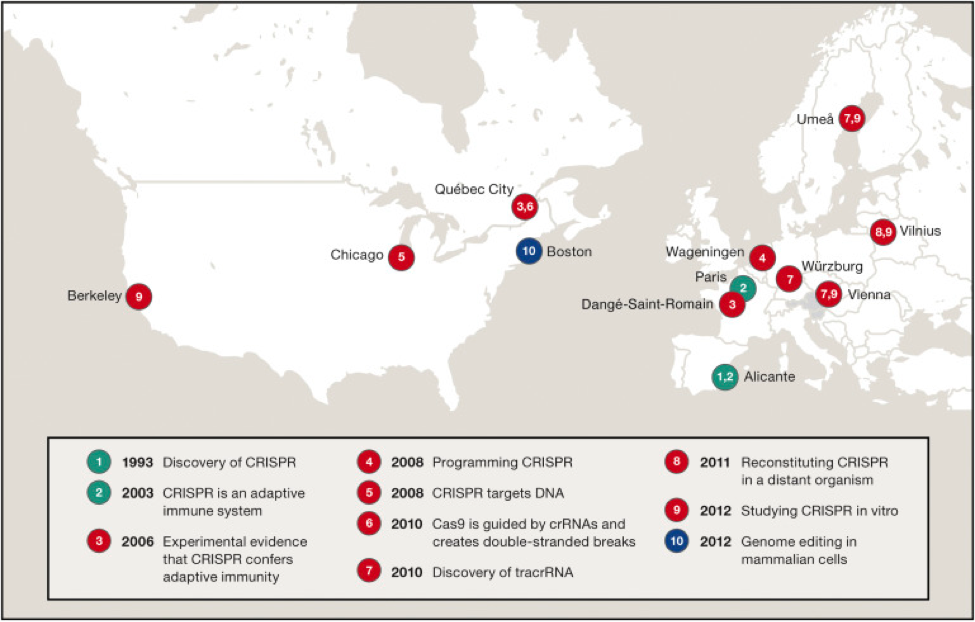
二十年來整個CRISPR系統的發展過程,訴説著許多不同的層面的故事,包含了科學的研究發現、發表論文所面對的困難、研究經費問題和此技術可能引發的科學倫理問題,以及社會大眾的認知。最初開研究動機只是想要了解耐鹽性微生物所含的奇怪重複序列、軍事上要抵抗生化武器或是食品工業上要增加優格的生產,並不是以編輯人類基因體或治療疾病為目的。而目前新興的科學研究方法,是在沒有假設前提下,去分析大數據及基因庫的資料。更特別的是,在CRISPR系統發展史上幾個關鍵發現的科學家都非常年輕,他們不懼風險願意挑戰,擁有強烈的研究動機與信心,卻常受限於沒有足夠的研究經費,這點暴露出科學研究經費資源分配問題,值得國家重新考量嚴肅面對。
參考文獻
- Lander, E.S. (2016). The Heroes of CRISPR. Cell 164, 18-28.
- Mojica, F.J., Juez, G., and Rodriguez-Valera, F. (1993). Transcription at different salinities of Haloferax mediterranei sequences adjacent to partially modified PstI sites. Molecular microbiology 9, 613-621.
- Mojica, F.J.M., Diez-Villasenor, C., Garcia-Martinez, J., and Soria, E. (2005). Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J Mol Evol 60, 174-182.
- Cong, L., Ran, F.A., Cox, D., Lin, S., Barretto, R., Habib, N., Hsu, P.D., Wu, X., Jiang, W., Marraffini, L.A., et al. (2013). Multiplex genome engineering using CRISPR/Cas systems. Science 339, 819-823.
- Mali, P., Yang, L., Esvelt, K.M., Aach, J., Guell, M., DiCarlo, J.E., Norville, J.E., and Church, G.M. (2013). RNA-guided human genome engineering via Cas9. Science 339, 823-826.
