聚合酶連鎖反應(PCR)發展的脈絡及歷程
聚合酶連鎖反應(Polymerase chain reaction, PCR)是一種簡單、廉價和可靠的複製DNA片段的方法,是目前分子生物學中最廣泛使用的技術,已被成功應用於生物醫學研究、犯罪取證、疾病診斷及分子考古學等領域 。PCR發展多年,技術逐漸成熟,並已演化出多種變體的PCR。我們將在本文中探索PCR發展的脈絡及歷程,並介紹三種世代的PCR(傳統擴增PCR、即時定量PCR及微滴式數字PCR)的作用、性質、特點及應用。
撰文/陳淵銓
●PCR的發展歷史
生物的DNA在活體外進行人工複製是一種耗時費事的程序,首先要將DNA經限制酶(restriction enzyme)裁剪,接者利用連接酶(ligase)加到適當的載體(vector)中,再利用轉形(transformation)的方法,如瞬間的電衝擊(electroporation)或熱休克(heat shock)等,將含有特定DNA片段的載體送到大腸桿菌勝任細胞(competent cell)中,再將此菌培養繁殖,經過分離及純化的步驟,才能大量複製DNA片段。
在1983年,凱利·穆利斯(Kary Mullis)開發PCR技術,並於1993年獲得諾貝爾化學獎,他利用DNA雙股複製的原理,使用DNA聚合酶用於體外試驗複製特定DNA片段,並在短期內大量擴增此片段。然而,在最初的PCR實驗中,穆利斯並非採用常溫來解開DNA雙鏈,而是把雙股DNA加熱到96℃,使其分離成為兩條DNA單鏈。在96℃下,因為大腸桿菌DNA聚合酶會被破壞,所以在每個循環的加熱步驟後均須補充新的酵素,並在整個過程中都需人工操作,造成PCR效率極低。
後來,研究人員改用自水生棲熱菌(Thermus aquaticus)分離出來的耐熱性DNA聚合酶(Taq DNA polymerase,最適作用溫度72 °C,由台灣科學家錢嘉韻於1976年在溫泉中發現)取代原本的大腸桿菌DNA聚合酶。由於Taq DNA聚合酶可耐高溫,故無需持續加入新的DNA聚合酶,使得反應過程變得簡單且可由機器操作,顯著提高了PCR的效率。然而,Taq DNA聚合酶所含的3'→5'核酸外切酶(exonuclease)活性缺乏校讀(proofreading)的能力,在DNA複製時容易發生錯誤,故又從其他細菌中獲得具有校讀功能的Pfu酶來降低突變率,Taq DNA聚合酶與Pfu酶的結合可以確保擴增DNA的準確性。
●PCR發展的三個世代
- 第一世代:傳統擴增PCR(traditional amplification PCR)
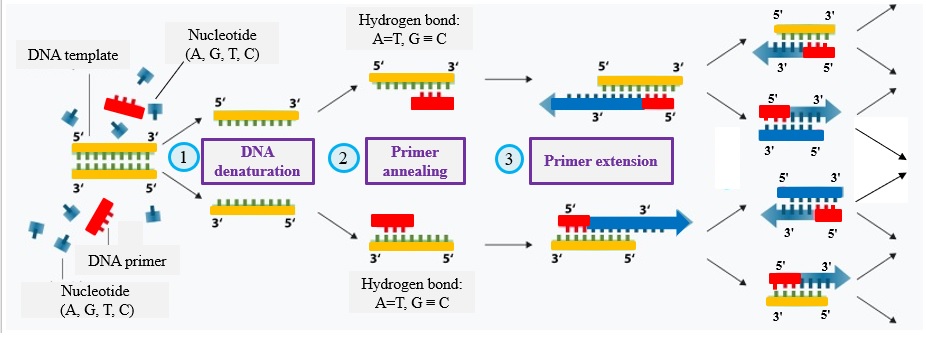
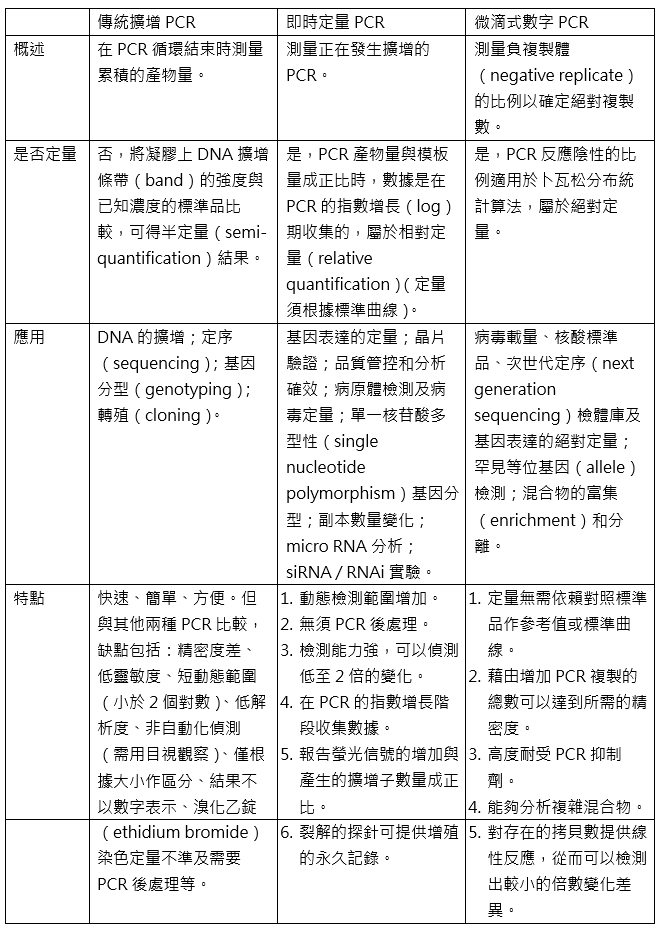
在試管內將特定DNA片段,以專一性的連鎖反應,使得DNA片段數目以2的對數(2n)快速增加而產生大量的產物。所需的材料包含DNA模板(template)、界定複製起始點兩端的引子(primer)、 Taq DNA聚合酶、 合成原料核苷酸(nucleotide: A, G, T, C)及緩衝溶液,關鍵步驟則是 DNA變性(denaturation)、引子黏合(annealing)及引子延長(extension)。
- 第二世代:即時定量PCR(quantitative real-time PCR)
用於即時檢測PCR產物的螢光染料標記在一段可與單股DNA模板進行專一性雜交的探針(如SyBR green或TaqMan probe)上,PCR儀的光學檢測系統記錄螢光信號強度,在某一循環中螢光信號的強度達到預先設定的閾值時,此時的循環數稱為Ct(threshold cycle)值或Cq(quantification cycle)值。Ct值與起始的DNA模板量成反比,起始的核酸量越多,達到閾值的循環數就越少,亦即Ct值會越小。以Ct值為縱坐標,起始模板數為橫坐標作出標準曲線,便可計算出待測DNA的複製數(copy number)。
- 第三世代:微滴式數字PCR(droplet digital PCR)
將待測的標的核酸分子均勻稀釋後,分散至大量的微反應器或微油滴中,並使每個反應只有0或1個DNA模板,經PCR擴增後,有1個DNA模板就會產生螢光訊號,而0個的則不會有螢光訊號,再採用卜瓦松分布(Poisson distribution)的統計模式,以分析軟體計算出標的核酸的絕對定量(absolute quantification)。
表1. 三個世代的 PCR 之比較
參考資料:
- Bartlett JMS, Stirling D. A Short History of the Polymerase Chain Reaction. PCR Protocols. Methods in Molecular Biology 226 2nd. 2003: 3–6. ISBN 1-59259-384-4. doi:10.1385/1-59259-384-4:3.
- Mullis KB, Erlich HA, Arnheim N, Horn GT, Saiki RK, Scharf SJ. Process for amplifying, detecting, and/or-cloning nucleic acid sequences. 1989 Jan. 24. 美國專利第4,683,195號 https://www.osti.gov/biblio/6254925-process-amplifying-detecting-cloning-nucleic-acid-sequences
- Hindson CM, Chevillet JR, Briggs HA, Gallichotte EN, Ruf IK, Hindson BJ, Vessella RL, Tewari Absolute quantification by droplet digital PCR versus analog real-time PCR. Nat Methods. 2013 Oct;10(10):1003-1005.doi: 10.1038/nmeth.2633.
- Poh TY, Nur Ali ABM, Chan LLY, Tiew PY, Chotirmall Evaluation of droplet digital polymerase chain reaction (ddPCR) for the Absolute Quantification of Aspergillus Species in the Human Airway. Int J Mol Sci. 2020 Apr 26;21(9):3043.doi: 10.3390/ijms21093043.