劍橋參考序列 (CRS)
劍橋參考序列 (Cambridge Reference Sequence,CRS)
國立臺灣大學醫學系呂明軒、臺北市立建國高中劉翠華教師
劍橋參考序列 (Cambridge Reference Sequence,CRS)是最廣為採用的人類粒線體 DNA 序列參考標準。
英國劍橋大學桑格博士 (Dr. Fred Sanger)是生物大分子研究領域的巨擘,發明了蛋白質與核酸的定序方法,於 1958 與 1980 年兩度獲得諾貝爾化學獎的殊榮。桑格博士的團隊於 1981 年首次發表人類粒線體 DNA (mt DNA) 全長約 $$16569$$ 個鹼基對,此序列稱為劍橋參考序列。
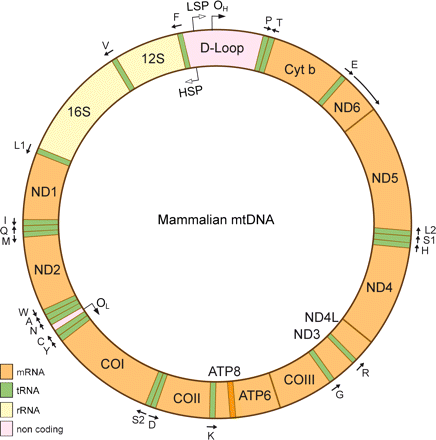
圖一 哺乳類粒線體DNA圖譜 橘色區域編碼訊息RNA (mRNA);綠色區域編碼轉運RNA (tRNA);黃色區域編碼核醣體RNA (rRNA);粉紅色區域為非編碼區。
圖片來源:http://jcb.rupress.org/content/193/5/809/F1.expansion.html
CRS 的發表揭開了粒線體這個擁有細胞核外 DNA 之胞器的神秘面紗。科學家們得以確定 $$29$$ 個已知基因在粒線體 DNA 上的相對位置,包含 12S 和 16S rRNA、$$22$$ 個 tRNA、$$3$$ 個細胞色素 c 氧化酶次單元、ATP 酶的次單元、及細胞色素 b(圖一),還從序列的特徵預測了 $$8$$ 個未知基因的存在。這些新發現的基因全都和氧化還原有關(包含 $$7$$ 個 NADH 去氫酶和另一個 ATP 酶的次單元)。
此外,科學家們發現,在粒線體所編碼的 $$37$$ 個基因之間,未編碼的區域極其稀少。絕大部分的生物粒線體基因缺乏內含子 (intron),相鄰基因往往沒有間隔的序列,甚至出現前一個基因的尾端與後一個基因的前端重疊的現象。基因尾端缺乏用來結束轉譯的終止密碼子(UAG、UAA、UGA)的情形亦不罕見。
這種序列的精簡程度,與細胞核內DNA中充滿未編碼序列的狀況相比,實在讓科學家們大開眼界。
隨著更多的粒線體 DNA 被定序,一些序列上的矛盾逐漸浮現。1999 年,科學家們發表了一篇修訂論文,指出 1981 年版本CRS中的 $$11$$ 個定序錯誤以及 $$7$$ 個罕見的鹼基變異。這次修訂的版本被特稱為 rCRS (revised CRS),也就是日後公認的標準劍橋參考序列。
CRS 不僅讓我們對粒線體 DNA 更加了解,也提供了演化親緣分析的一條途徑。比較不同粒線體 DNA 的序列可發現,特定位置的核苷酸會有不同的變異,此即單核苷酸多型性(single-nucleotide polymorphism,SNP)。
數個 SNP 若常常一起遺傳至下一代,則這些 SNP 的集合就是單倍體群(haplogroup)。科學家們研究大量的粒線體 DNA,將不同單倍體群加以分類,釐清了演化關係,同時也提供了人類遷移史的佐證(圖二)。值得注意的是,CRS 來自歐洲的單倍體群 H,是進行 SNP 比較時人為訂定的參考標準。由這些粒線體變異回溯可能的演化歷程,著名的粒線體夏娃(Mitochondrial Eve)才是單倍體群的共同祖先!
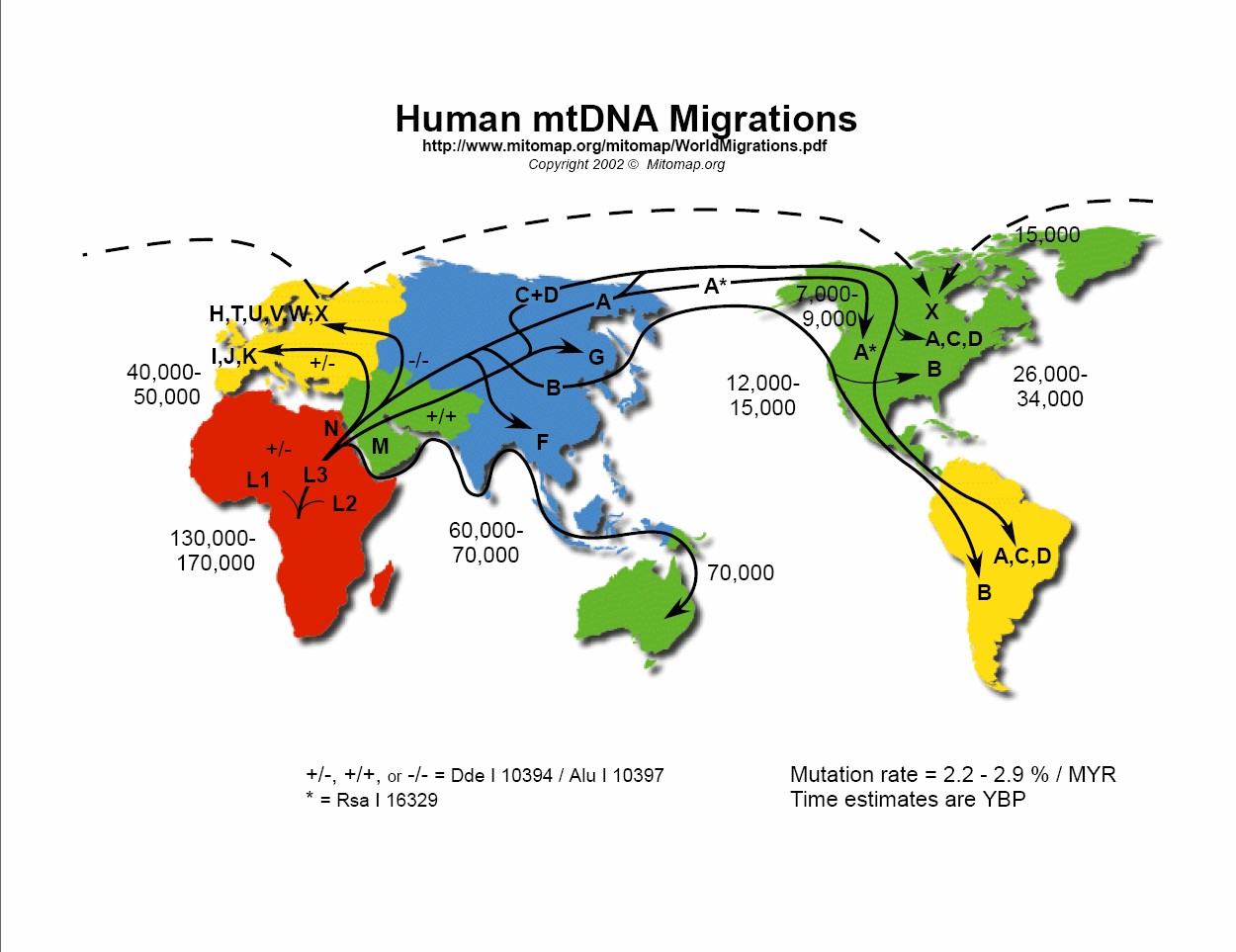
圖二 人類粒線體DNA遷徙過程 英文字母(部分帶有數字)代表單倍體群的分類群,其中非洲的L0為推測的單倍體群共同祖先。箭號表示推測的散布過程與變異,黑色數字表示單倍體群發生變異的推測距今年代。推估突變率為每一百萬年$$2.2$$~$$2.9\%$$。
圖片來源:http://www.jeffdonofrio.net/DNA/DNA_Analysis.html
參考資料
- Anderson, S. et al. (1981). Sequence and organization of the human mitochondrial genome. Nature 290 (5806): 457–465.
- Andrews, R. M. et al. (1999). Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA. Nat. Genet. 23 (2): 147.


 前一篇文章
前一篇文章 下一篇文章
下一篇文章 共軛焦顯微鏡的使用原理
共軛焦顯微鏡的使用原理 
